Umfassendes Bild der Zusammensetzung von Proteinverbünden unerlässlich
Die Mitochondrien der Bäckerhefe (Saccharomyces cerevisiae) enthalten rund 1.000 verschiedene Proteine. Für einige Proteinen wurde bereits gezeigt, dass sie ihre Funktion nur in festem Verbund mit anderen Proteinen, sogenannte Proteinkomplexen, erfüllen. Für die Mehrzahl der Proteine war hingegen wenig darüber bekannt, wie sie in Komplexen oder dynamischere Verbünden organisiert sind. Ein umfassendes Bild der Zusammensetzung dieser Proteinverbünde oder Proteinmaschinerien ist unerlässlich, um die molekularen Mechanismen und Wechselbeziehungen verstehen, die es den Mitochondrien ermöglichen, ihre vielfältigen biologischen Funktionen mit der bekannt hohen Präzision und Zuverlässigkeit zu erfüllen.
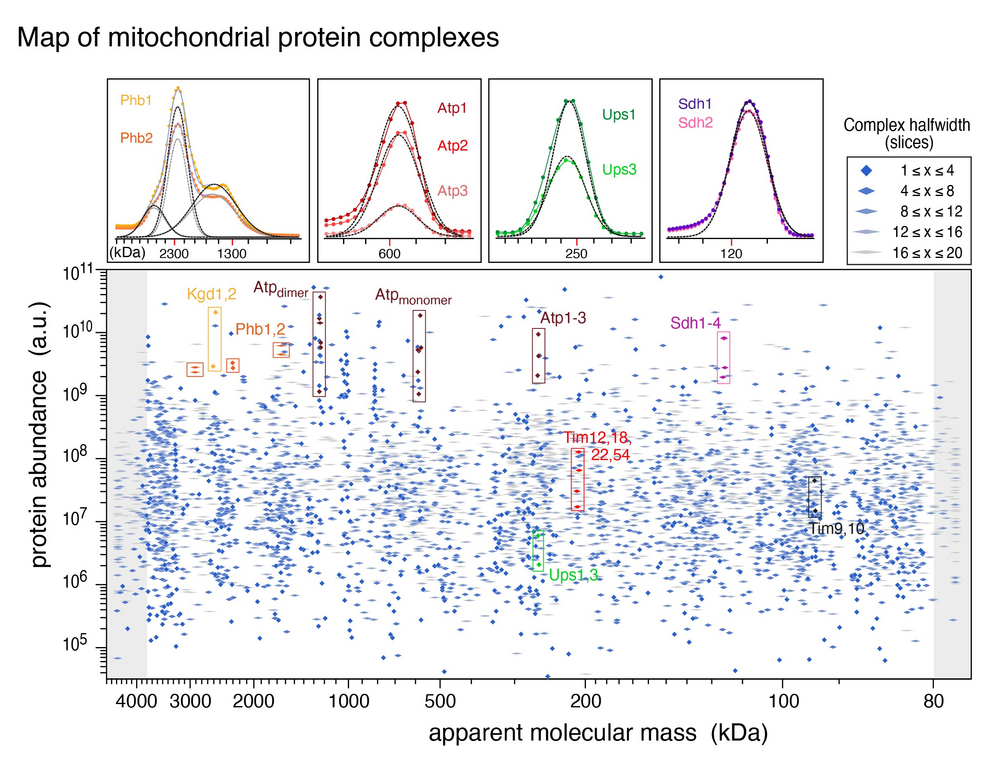
Für ihre Analysen setzten die Forschenden ein von ihnen entwickeltes Verfahren zur hochaufgelösten Komplexom-Analyse ein. Dabei werden Proteinkomplexe zunächst in intakter Form nach ihrer Größe in einem Gel aufgetrennt und dieses dann, nach Tieffrieren, in Scheibchen von 0.3 Millimeter Dicke geschnitten. Mittels Massenspektrometrie können dann alle in den Scheibchen enthaltenen Proteine identifiziert und mengen-analysiert werden. Die so erzeugten Protein-Profile, von den Forschenden zusammengenommen als MitCOM (für mitochondriales Complexom) bezeichnet, ergaben den bis dato umfassendsten und präzisesten Datensatz der quantitativen Größenverteilung von mehr als 90 Prozent der mitochondrialen Proteine. „Die Auswertung von MitCOM zeigte, dass mehr als 99% aller mitochondrialen Proteine in Komplexen, durchschnittlich sieben pro Protein, auftreten (zusammengenommen mehr als 5000 Signale von Proteinkomplexen, Abb. 1) organisiert sind“, sagte Dr. Uwe Schulte, der Erstautor der Arbeit. „Das ist ein deutlich höheres Maß an Komplexität als bislang angenommen wurde“. Dabei scheint die Komplexität nicht von den biochemischen Eigenschaften der einzelnen Proteine, sondern hauptsächlich von deren Funktion und Lokalisation in mitochondrialen Sub-Strukturen abzuhängen.
In einem zweiten Schritt nutzten die Forschenden die enorm große Dynamikbreite von MitCOM aus, um bislang unbekannte Verbindungen zwischen mehreren Signalwegen aufzuklären und neue Mechanismen zur Kontrolle des Proteinimports in Mitochondrien zu entschlüsseln. „MitCOM hat uns Wechselwirkungen von Proteinen mit dem TOM-Komplex gezeigt, die zusammen für die Qualitätskontrolle des Proteinimports verantwortlich sind“, erläutert Pfanner.
Die MitCOM-Daten stellen damit eine einzigartige Ressource dar, die sicherlich Anstoß zu weiteren spannenden Entdeckungen in der Mitochondrien-Forschung liefern wird. Dabei sind die Mitochondrien erst der Anfang. „Das Verfahren der Komplexom-Analyse lässt sich unmittelbar auf andere Organellen und Zellkompartimente übertragen und wird uns vermutlich noch viele weitere Einblicke in die evolutionäre Kreativität geben“, erklärt Fakler, der mit seinen Mitarbeiter*innen vor allem an der schnellen Signalübertragung an Zellmembrane arbeitet.
Original Pressemitteilung
Originalpublikation
Schulte, U., den Brave, F., Haupt, A., Gupta, A., Song, J., Müller, C.S., Engelke, J., Mishra, S., Mårtensson, C., Ellenrieder, L., Priesnitz, C., Straub, S.P., Doan, K.N., Kulawiak, B., Bildl, W., Rampelt, H., Wiedemann, N., Pfanner, N., Fakler, B., and Becker, T. (2023): Mitochondrial complexome reveals quality control pathways of protein import. In: Nature. https://doi.org/10.1038/s41586-022-05641-w
CIBSS Profil von Dr. Uwe Schulte
CIBSS Profil von Prof. Dr. Bernd Fakler
CIBSS Profil von Prof. Dr. Nikolaus Pfanner