Um natürliche Nukleasehemmer zu finden, hat CIBSS-Wissenschaftler Backofen vom Institut für Informatik an der Universität Freiburg mit seinem Team nun erstmals Deep Learning genutzt um ein Protein zu identifizieren, das die Aktivität von Cas13b blockiert. Deep Learning ist ein Teilbereich des Maschinellen Lernens und eignet sich dafür, große Datenmengen nach Mustern und Trends zu untersuchen. Ihre Ergebnisse stellen die Wissenschaftler*innen in der Zeitschrift Molecular Cell vor.
Auf der Suche nach Acrs: Erstmals Deep Learning angewandt
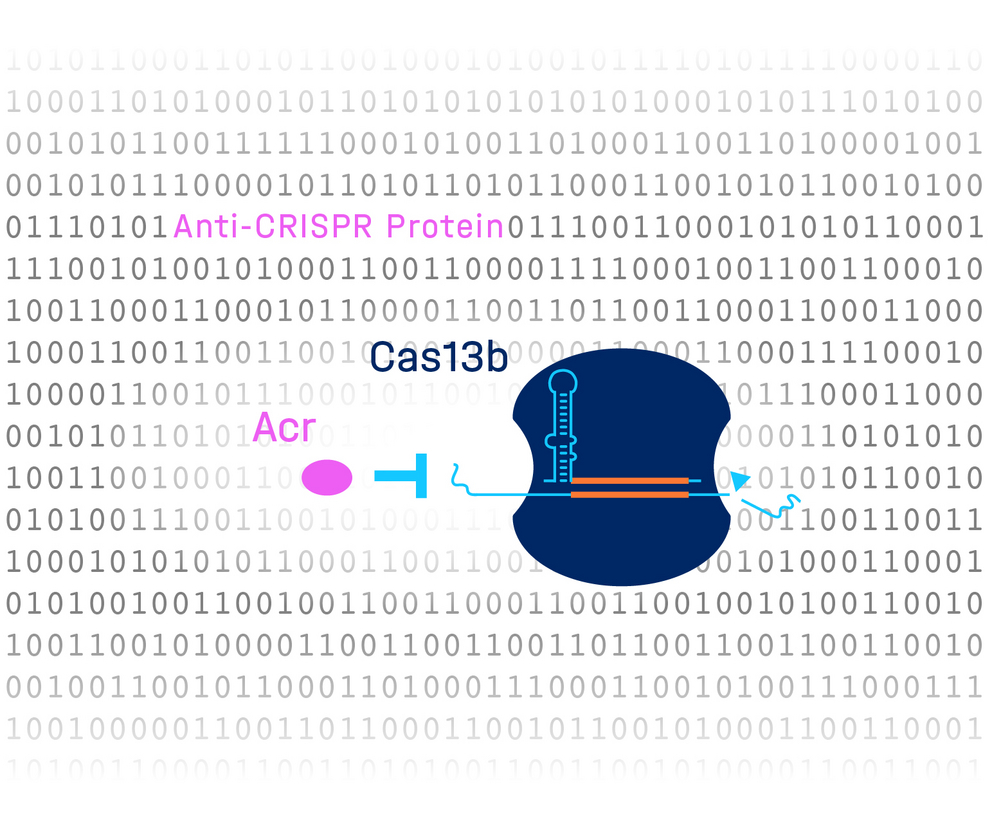
Wissenschaftler*innen vermuten, dass viele noch unentdeckte Proteine existieren, die Cas13b blockieren können – sogenannte Anti-CRISPR-Proteine (Arcs). Allerdings sind diese schwer zu finden. „Sie zu identifizieren heißt, eine Stecknadel im Heuhaufen zu finden, da Acrs einander überhaupt nicht ähneln“, erklärt Beisel, der in Kooperation mit Backofen federführender Wissenschaftler der Studie ist und die Leitung der Abteilung „Synthetische RNA-Biologie“ am Helmholtz-Institut für RNA-basierte Infektionsforschung in Würzburg innehat.
Deshalb haben die Mitglieder des Forschungsteams den Einsatz künstlicher Intelligenz vorangetrieben, um neue Acrs zu suchen. „Mit der Kombination aus unserem Deep Learning-Verfahren ‚DeepAcr‘ und dem Einsatz eines Hochdurchsatz-Screens ist es gelungen, das neue Anti-CRISPR-Protein zu entdecken“, sagt Backofen. „Die Millionen von Vorhersagen, die unser Algorithmus macht, helfen aber nicht nur, Anti-CRISPR Proteine zu finden. Der entwickelte Algorithmus zeigt, wie neuronale Netze auch bei komplexen Fragestellungen effektive Lösungsmöglichkeiten eröffnen"
Profil Rolf Backofen
Original Veröffentlichung
Wandera, K.G., Alkhnbashi, O.S., Bassett, H.V.I., Mitrofanov, A., Hauns, S., Migur, A., Backofen *, R., Beisel *, C.L. (2022): Anti-CRISPR prediction using deep learning reveals an inhibitor of Cas13b nucleases. In: Molecular Cell. DOI: https://doi.org/10.1016/j.molcel.2022.05.003 *:corresponding authors
Original-Pressemitteilung
https://kommunikation.uni-freiburg.de/pm/2022/deep-learning-hilft-gentherapien-und-antivirale-medikamente-zu-verbessern