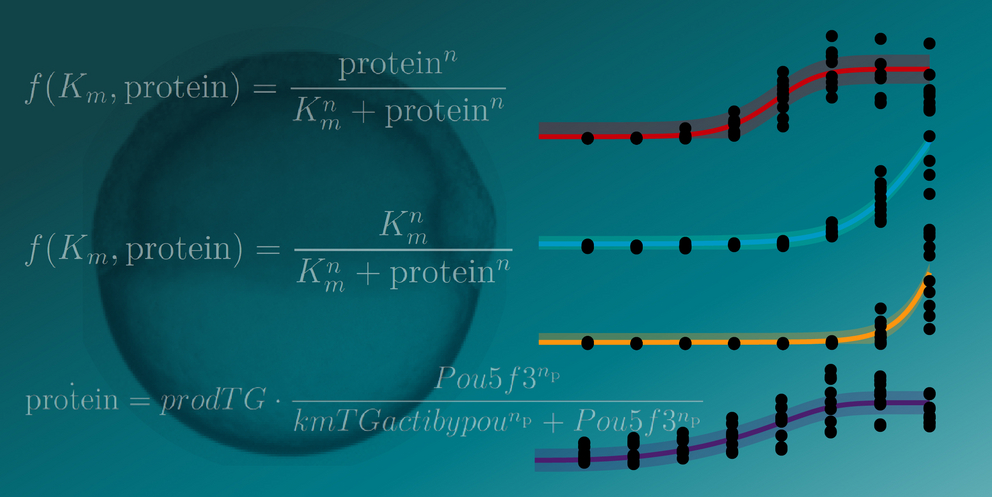Nach der Befruchtung der Eizelle müssen einige Gene sofort aktiviert werden, während andere noch inaktiv bleiben müssen. Zum Beispiel die Gene, die die Differenzierung von Zellen in spezialisierte Zelltypen auslösen, dürfen nicht zu früh aktiviert werden. Die aktuelle Studie unter Leitung der Entwicklungsbiologin Dr. Daria Onichtchouk und des theoretischen Physikers Prof. Dr. Jens Timmer beschreibt einen bisher unbekannten Mechanismus, durch den sich zwei aktivierende Transkriptionsfaktoren gegenseitig hemmen. Dieselben Faktoren, die die Expression früher Gene auslösen, können so auch die vorzeitige Expression von später Gene verhindern.
Die Forschenden machten diese Entdeckung an den Zebrafisch-Transkriptionsfaktoren Pou5f3 und Nanog. Die entsprechenden Moleküle bei Säugetieren, Oct4 und Nanog, sind medizinisch relevant, da sie die Bildung pluripotenter Stammzellen induzieren. Diese Technologie hat ein großes Potenzial für den zukünftigen Einsatz in der regenerativen Medizin und ihre Entdeckung wurde 2012 mit dem Nobelpreis ausgezeichnet.
“Wir wussten, dass diese Transkriptionsfaktoren miteinander interagieren, aber bisher ging man davon aus, dass sie die DNA immer nur zugänglicher und nicht weniger zugänglich machen können“, erklärt Onichtchouk. Die Forscher*innen konnten jetzt zeigen, dass es tatsächlich zwei mögliche Arten der Interaktion gibt: Pou5f3 und Nanog können synergistisch wirken, um ein Gen zu aktivieren, oder antagonistisch, um die Genexpression zu hemmen. „Wie sie interagieren, hängt von der Sequenz der DNA-Region ab, an die sie binden“, erklärt Onichtchouk. „Die neu entdeckte antagonistische Interaktion hemmt die Expression von Hunderten von Genen in frühen Entwicklungsstadien.“
Zu diesem Ergebnis sind die Forschungsgruppen von Onichtchouk und Timmer gekommen, indem sie ihre Expertise in Entwicklungsbiologie, Genetik und mathematischer Modellierung kombiniert haben. Beide sind Mitglieder des Exzellenzclusters CIBSS – Centre for Integrative Biological Signalling Studies an der Universität Freiburg, das solche interdisziplinären Projekte gezielt fördert.
Onichtchouks Arbeitsgruppe verglich zunächst mit genetischen Assays den Einfluss der Transkriptionsfaktoren auf die Expression von mehreren hundert Genen. Diese Daten gaben sie dann an Timmers Gruppe weiter. Sie bat sie, mögliche Regulationsmechanismen zu modellieren, ohne ihnen Anhaltspunkte dazu zu geben, welche Faktoren sie genau untersucht hatte.
„Die Formeln, die wir verwendet haben, waren tatsächlich sehr simpel und allgemeingültig, was den ganzen Ansatz so elegant und mich sehr glücklich macht“, sagt Jacques Hermes, der in der Gruppe von Timmer promoviert und zu dieser Studie einen großen Teil der Modellierungen beigetragen hat. Er fügt hinzu: „Sehr schön an dieser Studie ist auch, dass wir die Ergebnisse unseres Modells sofort anhand unabhängiger experimenteller Daten validieren konnten."
Das mathematische Modell und die experimentellen Daten zeigen, dass Pou5f3 und Nanog synergistisch diejenigen Gene aktivieren, bei denen sie beide als Aktivatoren wirken, aber eine antagonistische regulatorische Wirkung auf die Gene haben, die nur von einem der beiden aktiviert werden können. In ihrer Veröffentlichung vergleichen die Forschenden dieses Zusammenspiel mit zwei Schlüsseln und einem Schloss: Wenn einer der Schlüssel zwar in das Schloss passt, sich darin aber nicht drehen lässt, blockiert er den Zugang für den richtigen Schlüssel.
„Wir wissen jetzt viel mehr über die Mechanismen, durch die Transkriptionsfaktoren kontextabhängig funktionieren“, fasst Onichtchouk die Erkenntnisse zusammen. „Das macht unser Bild der Genregulation zwar noch komplexer, bringt uns aber auch einen wichtigen Schritt näher zu einem vollständigen Verständnis.“
Originalpublikation
Aileen Julia Riesle, Meijiang Gao, Marcus Rosenblatt, Jacques Hermes, Helge Hass, Anna Gebhard, Marina Veil, Björn Grüning, Jens Timmer, Daria Onichtchouk. Activator-blocker model of transcriptional regulation by pioneer-like factors. In: Nature Communications (2023). DOI: 10.1038/s41467-023-41507-z
Der Originalcode aus der Studie steht hier kostenlos zur Verfügung: https://github.com/vandensich/zebrafish-minimodels
CIBSS-Profil von Dr. Daria Onichtchouk
CIBSS-Profil von Prof. Dr. Jens Timmer
Die Studie wurde mit Mitteln der Deutschen Forschungsgemeinschaft (DFG) und des Bundesministeriums für Bildung und Forschung (BMBF) gefördert.